李艳课题组在《PNAS》发表论文-发现Gpr171通过ERK1/2与Notch信号通路调控胚胎造血干细胞的发生
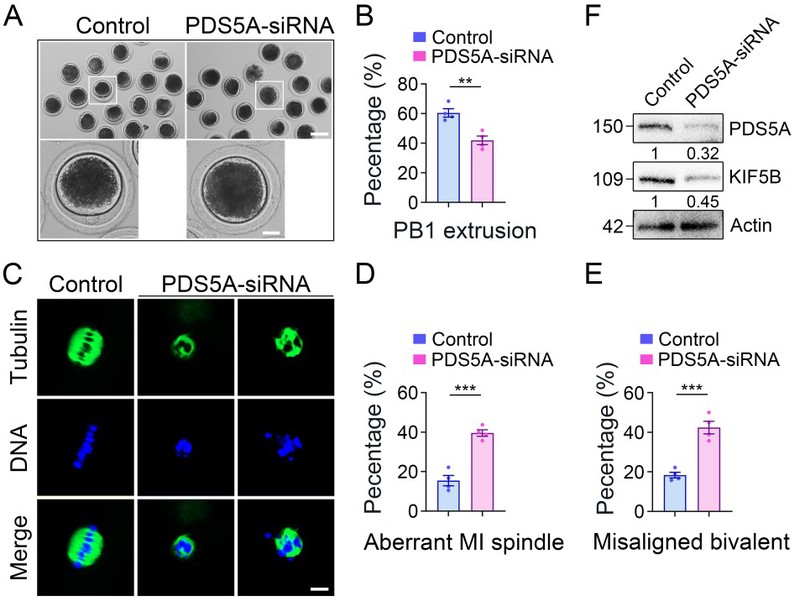
造血干细胞是维持人和动物终身血液供应的“生命种子”,对治疗白血病等恶性血液疾病至关重要。然而,如何在体外高效、稳定地制造出有功能的造血干细胞,一直是再生医学领域的一项重大挑战。深入研究造血干细胞在胚胎期的发育过程能有效指导体外培养获得造血干细胞,为血液疾病治疗带来新希望。2026年1月24日,浙江大学动物科学学院李艳研究员和医学院徐鹏飞研究员团队合作在国际著名学术期刊《美国科学院院刊》(PNAS)上在线发表题为“Gpr171 regulates embryonic hematopoietic stem cell emergence via ERK1/2 and Notch signaling”的研究论文。该研究发现G 蛋白偶联受体家族中的 GPR171是调控脊椎动物胚胎造血干细胞形成的关键因子,该受体与其配体协同作用,通过激活细胞内重要的 ERK1/2 和 Notch 信号通路,共同促进了造血干细胞的诞生。造血干细胞在胚胎发育时期起源于主动脉内皮细胞,通过内皮向造血转化过程产生。这一过程受到复杂信号网络和微环境的协同精细调控。然而,目前尚无法在体外完整复现该过程。该研究通过整合多平台高通量转录组数据,发现Gpr171基因在小鼠和斑马鱼胚胎造血干细胞发生过程中均呈现特异性高表达。通过构建Gpr171缺失的斑马鱼模型,发现Gpr171缺失会导致严重的胚胎造血干细胞缺陷。研究进一步揭示,GPR171 会与由 pcsk1nl 基因编码的内源性配体协同作用,通过独立且增效的方式激活 ERK1/2 与 Notch 两条信号通路,从而高效推动造血干细胞生成。此外,该调控机制在进化上具有保守性,Gpr171基因敲除的小鼠胚胎同样表现出功能性造血干细胞的显著减少。更令人振奋的是,当用其天然配体BigLEN(一种人体内也存在的小分子肽)处理斑马鱼胚胎时,造血干细胞的数量显著增加。这项突破性发现不仅首次揭示了GPR171 在造血干细胞起源中不可或缺的调控作用,加深了我们对生命起源的认识,更重要的是,它为未来在体外大规模生产功能性造血干细胞提供了全新的靶点和策略,为开发针对血液疾病和用于移植的干细胞疗法开辟了广阔的转化医学前景。浙江大学动物科学学院博士生周坤、刘宏艳和朱凯为本文共同第一作者,浙江大学动物科学学院李艳研究员、浙江大学医学院徐鹏飞研究员为本文共同通讯作者。这项工作得到了国家自然科学基金和杭州城西科创大走廊管委会资助。论文链接:https://www.pnas.org/doi/10.1073/pnas.2516989123(图文/李艳课题组 编辑/江丽军 初审/师福山 终审/任思丹) 科研与开发科 2026年1月24日