越来越多的研究表明,肠道微生物组与宿主基因组共同构成了“整体基因组”(holo-genome),宿主的复杂性状不仅受其基因组影响,也受肠道内微生物组以及二者之间相互作用的影响。因此,从整体基因组角度出发,能够为宿主复杂性状的遗传机制解析提供另一种思路。而整体基因组分析的前提是能够精准定义微生物分类、变异、功能等特征(microbial feature)在群体中的丰度与组成,这就需要利用一个完整的微生物基因目录来对这些微生物特征进行“按图索骥”。
猪的肠道中栖息着数十亿微生物,随着宏基因组学研究的深入,公共数据库中累积了大量的猪肠道宏基因组数据,使得我们依赖宏基因组组装基因组(MAG)的策略来进行微生物基因目录的构建成为了可能。此外,社区也需要一个可交互的MAG数据框使得猪整体基因组学研究更加便捷。
近日,浙江大学动物科学学院潘玉春教授/张哲副研究员团队在《NPJ Biofilms and Microbiomes》(IF5=9.9)上发表了题为《UPGG: expanding the taxonomic and functional diversity of the pig gut microbiome with an enhanced genome catalog》的研究论文。

(UPGG论文封面)
该研究整合了来自公共数据库及自测的5784份猪肠道宏基因组样本,构建了一个统一的猪肠道微生物基因组目录——UPGG,系统解析了猪肠道微生物的分类与功能多样性。该目录共鉴定出38,786个MAGs,其中24,802个为高质量MAGs,其数量与完整性均显著优于现有数据库。除此之外,目录中也囊括了超过7800万个基因和2300万个单核苷酸变异(SNVs),极大拓展了猪肠道微生物的遗传多样性。同时,UPGG也系统注释了抗生素抗性基因(ARGs)和代谢基因簇(MGCs),揭示了猪肠道菌群中巨大的功能潜力及潜在的耐药性风险。
研究还发现,宿主年龄、品种和地理因素是影响菌群组成的关键变量,其中中国地方猪种表现出更高的基因组独特性。饱和曲线分析表明,现有样本量已足以获得具有代表性的MAG。利用独立的真实宏基因组数据进行验证分析,发现与以往的基因目录相比,基于UPGG的微生物特征定义变得更加准确、全面。
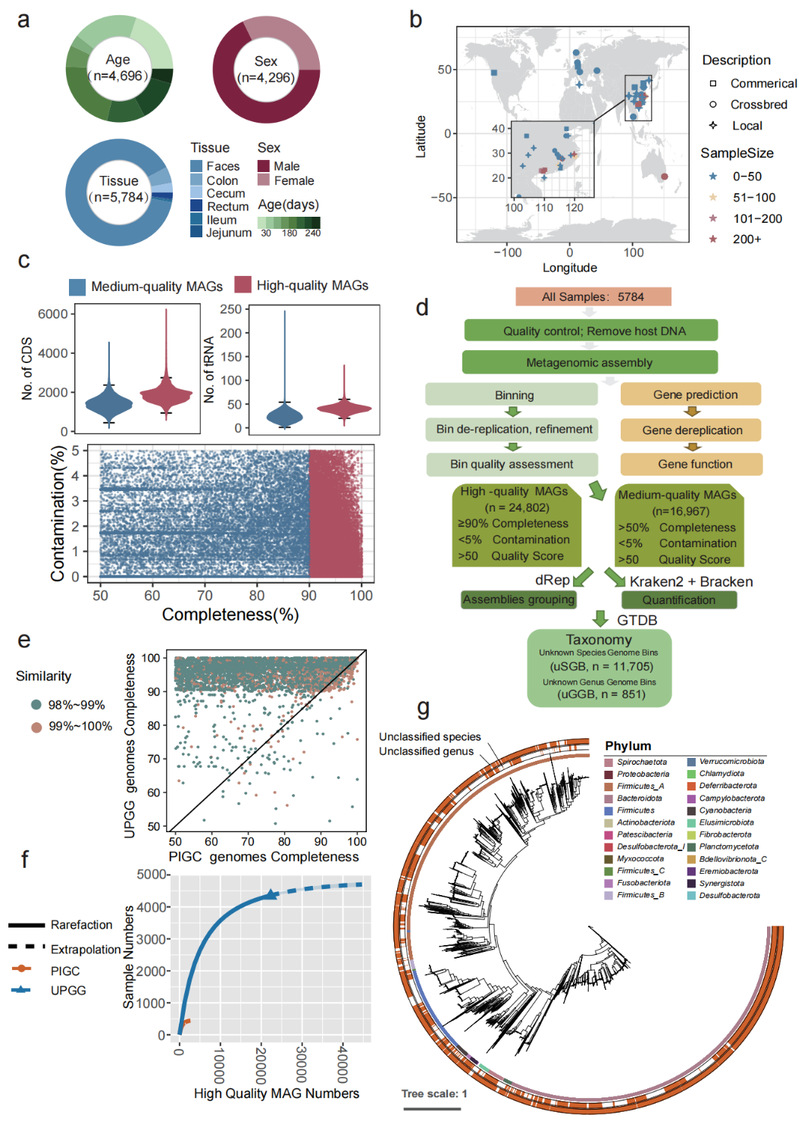
(UPGG构建主要流程图)
UPGG的建立不仅加深了我们对肠道菌群与宿主之间相互作用及共进化机制的理解,也有助于识别未被培养的微生物种类,从而深入分析微生物组如何随饮食、年龄、健康状况及环境条件变化。此外,鉴于猪与人类肠道菌群的高度相似性,UPGG还为人类健康相关研究提供了重要的模型和数据支持。
总体而言,该研究构建了一个专注于猪肠道微生物的统一基因组序列目录,通过对发育阶段、地理位置、品种和饲养方式的全面采样,显著拓展了猪肠道微生物组信息。另外,该研究提供了一个研究者可查看、检索、下载的在线网站(https://alphaindex.zju.edu.cn/upgg/#/),可以帮助科研人员进行基于宏基因组的微生物特征挖掘。
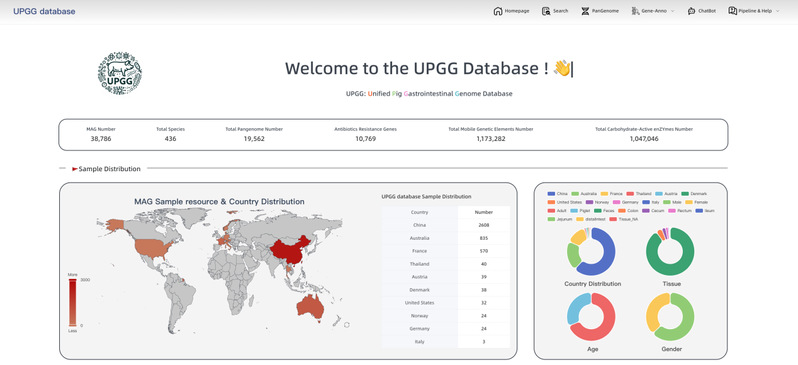
(UPGG网站主页)
该研究通过组装和分析猪肠道微生物的基因组,系统揭示了其潜在功能,为利用整体组学策略解析猪复杂性状遗传基础与开发微生物干预策略提供了较为坚实的数据基础。
浙江大学动物科学学院博士生刘爽和硕士生冯博为论文共同第一作者,浙江大学潘玉春教授和张哲副研究员为论文的共同通讯作者。本研究得到浙江省自然科学基金重点项目(LZ23C170003)、国家重点研发计划(2021YFD1200802、2023YFF1001100)的资助。
论文链接:https://doi.org/10.1038/s41522-025-00828-1
(图文/潘玉春课题组 编辑/江丽军 审核/师福山 终审/任思丹)
科研与开发科
2025年10月14日

